Extração de Diagnósticos e Informações de Doenças a partir de Registos Clínicos Eletrónicos: Uma Revisão Sistemática das Abordagens de Mineração de Texto: diferenças entre revisões
| Linha 139: | Linha 139: | ||
= Figuras e ilustrações = | = Figuras e ilustrações = | ||
[[Image:Resumo da pesquisa e seleção de artigos.png|500px| | [[Image:Resumo da pesquisa e seleção de artigos.png|500px| left| thumb | Figura 1. Resumo da pesquisa e seleção de artigos.]] | ||
= Tabelas = | = Tabelas = | ||
Revisão das 00h38min de 2 de março de 2016
|
Francisco Bischoff |
Marta Martins |
Miguel Duarte |
Resumo
Introdução: Com a disponibilidade dos registos clínicos eletrónicos, verificou-se a necessidade da mineração de texto e de dados usando diversos métodos. Este trabalho tem como objetivo uma revisão na literatura métodos mais utilizados neste domínio, e se possível uma comparação entre eles.
Métodos: Foi realizada uma pesquisa na literatura da PubMed utilizando as palavras chave “registo clínico eletrónico” e “mineração de texto”, sendo incluídos os artigos que utilizam a técnica de mineração de texto em registo clínicos eletrónicos, e excluídos aqueles que utilizaram apenas dados estruturados, não utilizavam aprendizagem de máquina ou não buscavam informações de doenças e diagnósticos.
Resultados: Os registos clínicos eletrónicos abrangem uma vasta gama de informação, verificando nos artigos extraídos que poucos deles satisfaziam os critérios de inclusão. Foi verificado que dos artigos incluídos, grande parte tinha como objetivo a identificação de Fenótipos Computacionais através de algoritmos como o de Campo Aleatório Condicional e Máquina de Vetores de Suporte.
Conclusões: Desde o início da utilização dos registos clínicos eletrónicos a mineração de texto tem demonstrado grande potencial. Revisões anteriores tiveram um enfoque alargado sendo que esta revisão se focou na mineração de texto. A quantidade de estudos nesta área está por crescer, sendo necessária a realização de meta-análise para comparar as suas performances de forma mais ampla e significativa.
Palavras-chave: mineração de texto, mineração de dados, algoritmos, diagnósticos, informações de doenças, registos clínicos eletrónicos
Introdução
As últimas décadas foram marcadas por uma crescente disponibilidade de registos clínicos eletrónicos (RCE). Além disso, técnicas de mineração de texto e mineração de dados têm sido intensamente estudadas como uma ferramenta para a descoberta de conhecimento. Várias dessas técnicas têm sido aplicadas para extrair ou interligar as informações de RCE, a fim de melhorar os sistemas de apoio à decisão, encontrar novos padrões ou conhecimento até então desconhecidos. Cada sistema de mineração de texto tem diferentes implementações e metas, por exemplo associações doença-doença, doença-droga, doença-genes, e alguns podem ser melhores para um objetivo, mas não para outros. Compreender as diferentes abordagens para diferentes objetivos é fundamental para garantir os melhores resultados.
O principal objetivo deste estudo é a identificação dos métodos de mineração de texto utilizados na extração de informação sobre doenças e diagnósticos a partir dos RCEs. Os objetivos secundários são comparar as medidas de desempenho destas abordagens.
Métodos
Critérios de inclusão e exclusão
Foram selecionados estudos que comparam o uso de uma técnica de mineração de texto em RCE com um padrão de referência. Foram excluídos estudos que utilizam somente dados estruturados ou semiestruturados (ex.: resultados de laboratórios, documentos previamente anotados), estudos que não utilizam métodos de machine learning e estudos que não tentam extrair informações sobre doenças ou diagnósticos e co-morbilidades. Estudos realizados em idiomas para além do inglês, alemão, espanhol e português não foram considerados.
Fontes e estratégias de busca
A estratégia de busca na literatura foi desenvolvida utilizando o Medical Subject Headings (MeSH) e palavras de texto relacionadas com RCE, mineração de texto e mineração de dados.
A pesquisa não foi limitada por data ou idioma. Um exemplo da consulta efetuada no PUBMED é descrito como se segue: ("text mining"[All Fields] AND ("electronic health records"[MeSH Terms] OR "electronic health records"[All Fields] OR "electronic health record"[All Fields])).
Processo de seleção e obtenção de dados
Os resultados da pesquisa foram inicialmente geridos através do Mendeley, um software gestor de referências, onde foram removidos os duplicados, e depois enviados para o Covidence, uma plataforma online que facilita a colaboração entre os revisores durante os processos de seleção dos estudos, avaliação da qualidade e extração de dados. Os autores desenvolveram e testaram perguntas de triagem e formulários para as avaliações dos artigos com base nos critérios de inclusão e exclusão, tendo sido feito um exercício de calibração para aperfeiçoar as perguntas de triagem.
Os autores analisaram de forma independente os títulos e resumos. Pares de autores analisaram os textos completos dos artigos pré-selecionados. As divergências foram decididas através da discussão, sendo a opinião do terceiro autor utilizada como desempate. Os nomes das revistas, autores e instituições não foram ocultados dos revisores no momento da seleção.
Usando um formulário padronizado no software Covidence, ambos os autores extraíram os dados de forma independente e em duplicado de cada estudo. Para garantir a consistência entre os revisores, foram realizados exercícios de calibração antes de iniciar a avaliação. Os autores resolveram as divergências pela discussão, tendo uma estatística κ de 0,726.
Foram extraídos os seguintes dados dos estudos:
- Fonte dos dados (ex.: notas de admissão, notas de alta);
- Quantidade de dados utilizados nos treinos e testes (ex.: número de episódios de internamento, número de registos dos pacientes);
- Método de validação (ex.: validação cruzada, corpora independentes);
- Algoritmo de Machine Learning (ex.: Naïve Bayes, K-NN; SVM);
- Características de mineração (ex.: processamento de linguagem natural, expressões regulares, saco de palavras)
- Objetivos dos processos de mineração (ex.: fenotipagem, classificação);
- Dados da tabela 2x2, se disponíveis (ex.: verdadeiros positivos, verdadeiros negativos).
- Medidas de desempenho, se disponíveis (ex.: precisão, recall, F1 score).
Resultados
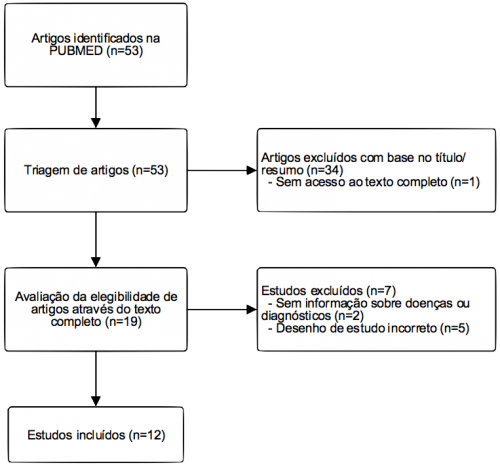
Os resultados da pesquisa de artigos estão apresentados na Figura 1. Dos 53 estudos iniciais, 12 artigos enquadraram nos critérios de inclusão. Todos os artigos forneceram dados suficientes para extrair as medidas de desempenho.
Risco de viés nos estudos incluídos
A avaliação de risco de viés dos 12 estudos incluídos indicam um alto risco de viés em 3 artigos (25%), risco incerto em 3 artigos (25%) e baixo risco em 6 artigos (50%). Os itens de avaliação de risco de viés estão resumidos na Tabela 1.
Métodos de mineração de texto
Nos estudos selecionados, verifica-se que a grande maioria tem como objetivo ou utiliza numa fase intermédia a identificação de Fenótipos Computacionais (ou Definição de Fenótipos através de RCE), os quais se definem como uma condição ou característica clínica, ou um conjunto de atributos clínicos que podem ser determinados através dos dados do RCE e dados auxiliares sem a necessidade de uma revisão ou interpretação por um clínico[1]. Foram agrupados para fins de análise estudos que utilizavam estes métodos num grupo chamado Fenotipagem.
Alnazzawi et al. utilizaram o processamento de linguagem natural (PLN) com a finalidade de construir um corpus de fenótipos para a identificação de insuficiência cardíaca, publicamente disponível online (http://www.nactem.ac.uk/PhenoCHF/)[2]. Informações da sintaxe[2][3] [4] [5], afixos de palavras[2] e etiquetadores morfológicos[2][5][6] foram algumas das técnicas utilizadas por alguns dos artigos. Byrd et al. verificou no seu trabalho que a maioria dos critérios de diagnósticos para insuficiência cardíaca eram constituídos por substantivos compostos sendo um ponto importante a identificação de frases que contêm tais substantivos[3].
Geralmente após a anotação do texto através do PLN, é realizada uma análise do texto. Byrd et al. utilizou diversas técnicas como coocorrência, desambiguidade, negação, contrafatual, restrições numéricas e de segmento[3]. Giang et al. utilizou a técnica chamada Similaridade Semântica sob o pressuposto de que duas palavras ou termos que coexistam na mesma frase ou documento possuem um contexto similar, sendo possível assim deduzir a sua distância semântica[4][7].
O uso do saco de palavras, embora tenha muitas limitações como a perda da ordem dos elementos e de informações de contexto, ainda tem sido bastante utilizado pela sua simplicidade[2][5][6][8]. Schuemie et al. além do saco de palavras, utilizou n-gramas como forma de mitigar algumas destas limitações[8].
Como forma de redução da dimensionalidade e do “ruído” dos documentos, verifica-se a aplicação de técnicas como remoção de stop words[9] [10] [11], de palavras que aparecem apenas uma vez[9][10], de palavras com menos de 3 letras[9][10], stemming[11] e associação de palavras[8].
Gerdes et al.[12] utiliza um software proprietário chamado SAS® Text Miner de forma a realizar PLN, entretanto não fornece mais informações. Enquanto DeShazo et al. utiliza um corpus anotado manualmente[13] [14].
Uso de terminologias padronizadas
As técnicas de mineração de texto são muito dependentes dos léxicos, terminologias (ou vocabulário) e ontologias. Um léxico pode ser útil para o mapeamento de conceitos, entretanto as terminologias agrupam termos de um particular domínio de interesse, abordando problemas de sinonímia, polissemia e desambiguidade. As ontologias podem ser representadas de diversas formas, incluindo classes, conceitos e tipos, ligados por relações e propriedades[14]. Dentre os artigos selecionados, alguns utilizam dicionários direcionados ou personalizados[3][4][5][6][15] enquanto outros utilizam terminologias padronizadas como o Unified Medical Language System (UMLS)[5][8][9][10][16].
Algoritmos de aprendizagem de máquina
Uma grande variedade de algoritmos tem sido utilizada como está resumido na Tabela 2.
Dos artigos que tem como objetivo a fenotipagem, os algoritmos de Campo Aleatório Condicional (CAC, ou CRF para Conditional Random Field) e Árvore de Decisão demonstraram um bom desempenho no F1 score[2][3][5][11]. Também se verifica um bom desempenho com a Máquina de Vetores de Suporte (MVS ou SVM para Support Vector Machine)[4][6][13] e Similaridade Semântica.
Os restantes artigos[9][16][17] têm como objetivo a classificação em duas categorias, exceto o artigo de Schuemie et al.[8] cujo objetivo era atribuir códigos ICD-9-CM presentes em relatórios de radiologia.
Na Tabela 3 estão resumidas as medidas de performance dos artigos de acordo com os objetivos e algoritmos utilizados.
Discussão
A conversão de dados não estruturados para dados estruturados é um dos passos mais importantes para a análise e geração de conhecimento. Verifica-se que existem diversas abordagens para tratar esta questão, sendo que a mais apropriada depende de diversos fatores individuais de cada fonte de dados e o objetivo da mineração de dados. Verifica-se também que os algoritmos mais utilizados foram o CAC e a MVS.
A comparação do desempenho dos algoritmos entre os artigos é dificultada pela heterogeneidade dos métodos de mineração de texto, o que já era esperado. Apenas os artigos de McCart et al.[10] e Schuemie et al.[8] demonstraram a utilização de diversos algoritmos utilizando o mesmo método de mineração de texto. Podemos evidenciar que a simples alteração do objetivo da mineração de texto influencia no desempenho dos algoritmos.
Conclusão
Desde o início da utilização dos RCEs a mineração de texto tem demonstrado um grande potencial para melhorar os cuidados de saúde. Nesta revisão pudemos verificar as diferentes metodologias utilizadas para em RCEs para obtenção de conhecimento, com enfoque em mineração de texto. Revisões anteriores[18] tiveram um enfoque alargado sendo também notável a heterogeneidade das abordagens utilizadas. A quantidade de estudos no âmbito desta revisão ainda está por crescer, sendo necessária futuramente a realização de estudos de meta-análise comparando suas performances de forma mais ampla e significativa.
Declarações
Lista de abreviaturas
- RCE – Registo Clínico Eletrónico
- MeSH – Medical Subject Headings
- PLN – Processamento de Linguagem Natural
- UMLS – Unified Medical Language System
- CAC – Campo Aleatório Condicional
- CRF – Conditional Random Field
- MVS – Máquina de Vetores de Suporte
- SVM – Support Vector Machine
Consentimento para publicação
Não aplicável.
Conflito de interesses
Os autores declaram a ausência de conflito de interesses.
Financiamento
Este estudo foi apoiado pela Universidade do Porto, que possibilitou a obtenção dos artigos de texto completo através da sua plataforma online.
Contribuição dos autores
Todos os autores participaram na pesquisa, análise e elaboração desta revisão.
Figuras e ilustrações
Tabelas
Tabela 1 – Resumo da avaliação de risco de viés conforme o QUADAS-2
![]() = baixo risco;
= baixo risco; ![]() = alto risco;
= alto risco; ![]() = risco incerto
= risco incerto
Tabela 2 – Resumo dos algoritmos utilizados nos estudos
| Algoritmo de Aprendizagem de Máquina | Estudos |
|---|---|
| Árvores de decisão | [3][8] |
| Campo aleatório condicional | [2][5][11] |
| k-NN | [8] |
| Regressão logística | [10] |
| Naïve Bayes | [8] |
| Random Forest | [8] |
| RIPPER | [8] |
| Máquina de vetores de suporte | [6][8][9][10][13] |
Tabela 3 – Resumo das performances
| Artigo | Objetivo | Algoritmo | N Teste | Precisão | Recall | F-1 Score |
|---|---|---|---|---|---|---|
| Alnazzawi et al.[2] | Fenotipagem | CAC† | 310 | 0,890 | 0,830 | 0,859 |
| Hammond et al.[11] | 200 | 0,816 | 0,884 | 0,848 | ||
| Jonnagaddala et al.[5] | 514 | 0,814 | 0,847 | 0,830 | ||
| Byrd et al.[3] | Árvore de decisão | 400 | 0,881 | 0,896 | 0,889 | |
| DeShazo et al.[13] | MVS‡ | 300 | 0,952 | 0,952 | 0,952 | |
| Patel et al.[6] | 7678 | 0,903 | 0,807 | 0,852 | ||
| Gerdes et al.[12] | SAS® CCS | 200 | 0,560 | 0,700 | 0,622 | |
| Giang et al.[4] | Similaridade semântica | 20 | 0,857 | 1,000 | 0,923 | |
| Luther et al.[9] | Classificação | MVS‡ | 7356 | 0,825 | 0,921 | 0,870 |
| Tanushi et al.[16] | [Dicionário]* | 1867 | 0,977 | 0,603 | 0,746 | |
| McCart et al.[10] | Regressão logística | 2846 | 0,852 | 0,846 | 0,849 | |
| MVS‡ | 0,860 | 0,840 | 0,850 | |||
| MVS-Cost (MetaCost) | 0,787 | 0,931 | 0,853 | |||
| Schuemie et al.[8] | MyC | 974 | 0,850 | 0,900 | 0,874 | |
| RIPPER | 0,870 | 0,920 | 0,894 | |||
| C4.5 | 0,900 | 0,930 | 0,915 | |||
| MVS‡ | 0,870 | 0,910 | 0,890 | |||
| Random Forest | 0,820 | 0,860 | 0,840 | |||
| k-NN | 0,960 | 0,590 | 0,731 | |||
| Naïve Bayes | 0,720 | 0,920 | 0,808 | |||
| Atribuição de diagnósticos ICD-9-CM | MyC | 976 | 0,900 | 0,840 | 0,869 | |
| RIPPER | 0,880 | 0,850 | 0,865 | |||
| C4.5 | 0,880 | 0,840 | 0,860 | |||
| MVS‡ | 0,840 | 0,820 | 0,830 | |||
| Random Forest | 0,890 | 0,630 | 0,738 | |||
| k-NN | 0,840 | 0,550 | 0,665 | |||
| Naïve Bayes | 0,560 | 0,690 | 0,618 |
† Campo Aleatório Condicional, ‡ Máquina de Vetores de Suporte, *uso apenas de dicionário para identificação da presença de um termo
Referências
- ↑ Rachel R, Michelle S. Electronic Health Records-Based Phenotyping. Rethinking Clinical Trials. [1]. Published 2014. Accessed February 21, 2016
- ↑ 2,0 2,1 2,2 2,3 2,4 2,5 2,6 2,7 Alnazzawi N, Thompson P, Batista-Navarro R, Ananiadou S. Using text mining techniques to extract phenotypic information from the PhenoCHF corpus. BMC Med Inform Decis Mak. 2015;15 Suppl 2(Suppl 2):S3. [doi:10.1186/1472-6947-15-S2-S3]
- ↑ 3,0 3,1 3,2 3,3 3,4 3,5 3,6 Byrd RJ, Steinhubl SR, Sun J, Ebadollahi S, Stewart WF. Automatic identification of heart failure diagnostic criteria, using text analysis of clinical notes from electronic health records. Int J Med Inform. 2014;83(12):983-992. [doi:10.1016/j.ijmedinf.2012.12.005]
- ↑ 4,0 4,1 4,2 4,3 4,4 Giang P, Williams A, Argyros L. Automated extraction of the Barthel Index from clinical texts. AMIA Annu Symp Proc. 2013;2013(Clc):486-495. [2][http: //www.pubmedcentral.nih.gov/articlerender.fcgi?artid= PMC3900193]
- ↑ 5,0 5,1 5,2 5,3 5,4 5,5 5,6 5,7 Jonnagaddala J, Liaw S-T, Ray P, Kumar M, Dai H-J, Hsu C- Y. Identification and Progression of Heart Disease Risk Factors in Diabetic Patients from Longitudinal Electronic Health Records. Biomed Res Int. 2015;2015:636371. [doi:10.1155/2015/636371]
- ↑ 6,0 6,1 6,2 6,3 6,4 6,5 Patel R, Jayatilleke N, Broadbent M, et al. Negative symptoms in schizophrenia: a study in a large clinical sample of patients using a novel automated method. BMJ Open. 2015;5(9):e007619. [doi:10.1136/bmjopen-2015-007619]
- ↑ Budanitsky A, Hirst G. Semantic distance in WordNet : An experimental , application-oriented evaluation of five measures. Work WordNet Other Lex Resour Second Meet North Am Chapter Assoc Comput Linguist. 2001;2(12):29- 34. [doi:10.1.1.29.2985]
- ↑ 8,00 8,01 8,02 8,03 8,04 8,05 8,06 8,07 8,08 8,09 8,10 8,11 8,12 Schuemie MJ, Sen E, ’t Jong GW, van Soest EM, Sturkenboom MC, Kors JA. Automating classification of free-text electronic health records for epidemiological studies. Pharmacoepidemiol Drug Saf. 2012;21(6):651- 658. [doi:10.1002/pds.3205]
- ↑ 9,0 9,1 9,2 9,3 9,4 9,5 9,6 Luther SL, McCart JA, Berndt DJ, et al. Improving identification of fall-related injuries in ambulatory care using statistical text mining. Am J Public Health. 2015;105(6):1168-1173. [doi:10.2105/AJPH.2014.302440]
- ↑ 10,0 10,1 10,2 10,3 10,4 10,5 10,6 10,7 McCart JA, Berndt DJ, Jarman J, Finch DK, Luther SL. Finding falls in ambulatory care clinical documents using statistical text mining. J Am Med Inform Assoc. 2013;20(5):906-914. [doi:10.1136/amiajnl-2012-001334]
- ↑ 11,0 11,1 11,2 11,3 11,4 Hammond KW, Ben-Ari AY, Laundry RJ, Boyko EJ, Samore MH. The Feasibility of Using Large-Scale Text Mining to Detect Adverse Childhood Experiences in a VA-Treated Population. J Trauma Stress. 2015;28(6):505-514. [doi:10.1002/jts.22058]
- ↑ 12,0 12,1 Gerdes LU, Hardahl C. Text mining electronic health records to identify hospital adverse events. In: Studies in Health Technology and Informatics. Vol 192. Netherlands; 2013:1145. [doi:10.3233/978-1-61499-289-9-1145]
- ↑ 13,0 13,1 13,2 13,3 DeShazo JP, Turner AM. An interactive and user-centered computer system to predict physician’s disease judgments in discharge summaries. J Biomed Inform. 2010;43(2):218-223. [doi:10.1016/j.jbi.2009.08.016]
- ↑ 14,0 14,1 Kumar VD, Tipney HJ. Biomedical Literature Mining. Vol 1159. (Kumar VD, Tipney HJ, eds.). New York, NY: Springer New York; 2007. [doi:10.1109/BIBE.2007.4375765]
- ↑ Ehrentraut C, Tanushi H, Dalianis H, Tiedmann J. Detection of Hospital Acquired Infections in sparse and noisy Swedish patient records.: A machine learning approach using Naive Bayes, Support Vector Machines and C4. 5. Sixth Work Anal Noisy Unstructured Text Data, AND, held conjunction with Coling 2012. 2012
- ↑ 16,0 16,1 16,2 Tanushi H, Kvist M, Sparrelid E. Detection of healthcare- associated urinary tract infection in Swedish electronic health records. Stud Health Technol Inform. 2014;207:330-339. [doi:10.3233/978-1-61499-474-9-330]
- ↑ Berndt DJ, McCart JA, Finch DK, Luther SL. A Case Study of Data Quality in Text Mining Clinical Progress Notes. ACM Trans Manag Inf Syst. 2015;6(1):1:1-1:21. [doi:10.1145/2669368]
- ↑ Iavindrasana J, Cohen G, Depeursinge A, Müller H, Meyer R, Geissbuhler A. Clinical data mining: a review. Yearb Med Inform. 2009;(December 2015):121-133. [doi:me09010121] pii